
레 꾸인 마이 박사가 이끌고 베트남 국립대학교 하노이 캠퍼스 자연과학 대학의 지원을 받은 "생명공학을 이용한 미량 영양소 함량 개선 벼 품종 개량" 연구 프로젝트는 벼의 미량 영양소 함량을 향상시키고 건강에 해로운 중금속 축적 위험을 최소화하는 데 상당한 성과를 거두었습니다.
식단 내 미량 영양소 결핍 문제와 농산물 카드뮴 오염에 대한 우려가 커짐에 따라, 연구팀은 CRISPR/Cas9 유전자 편집 기술을 활용한 첨단 접근법을 통해 곡물 내 철과 아연 축적을 증가시키고 카드뮴 흡수를 감소시키며 식물 생장을 유지하는 방법을 모색했습니다. 본 프로젝트의 목표는 유전자 편집 구조를 설계하고, 재생 과정을 확립하여, 기존 품종 대비 철과 아연 함량이 증가하고 카드뮴 함량이 최소 10% 감소된 개량 벼 품종을 개발하는 것이었습니다.
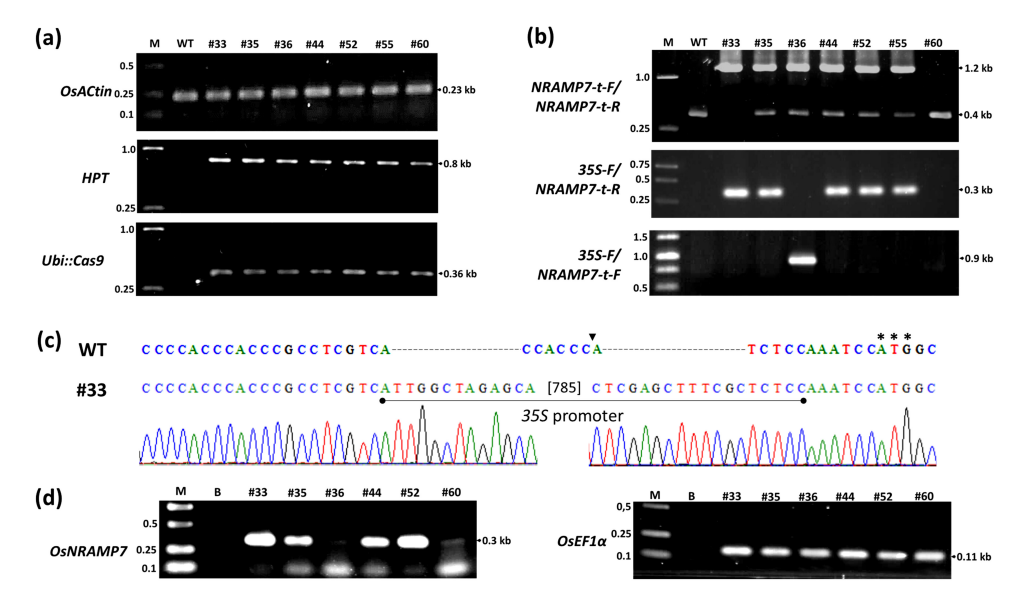
연구팀은 구현 과정에서 철, 아연, 카드뮴 등 벼의 금속 수송 유전자 OsNRAMP2, OsNRAMP5, OsNRAMP7을 표적으로 하는 4개의 CRISPR/Cas9 벡터와 OsYSL2 유전자 발현을 조절하는 pCRISPRa 벡터를 성공적으로 설계 및 검증했습니다. 이들 유전자는 벼에서 철, 아연, 카드뮴의 수송 및 축적에 중요한 역할을 합니다. 유전자 편집 및 식물체 재생 과정은 유전자 전달 효율과 돌연변이 효과에 대한 기준을 충족하도록 개발되었으며, 이를 통해 재생 잠재력이 높은 품종에 적용할 수 있게 되었습니다. 이러한 기술적 기반을 바탕으로 연구팀은 R7-33.47, R5-01.59, R5-04.28, R5-22.21의 네 가지 유전자 편집 벼 계통을 개발했습니다. 북방산악농림과학기술연구소의 독립적인 분석 결과, 이들 벼 종자의 철과 아연 함량은 원품종 대비 최소 10% 증가했고, 카드뮴 함량은 최소 10% 감소한 것으로 확인되었습니다.
연구팀은 유망한 벼 품종 개발에 성공했을 뿐만 아니라, 베트남에서 널리 재배되는 여러 벼 품종의 철, 아연, 카드뮴 함량을 평가한 보고서와 벼의 미량 영양소 관련 유전자 편집 공정을 포함한 포괄적인 과학 기술 성과를 달성했습니다. 특히, 이 공정은 캘러스 조직 총 개수를 기준으로 10% 이상의 재생 효율, 재생된 식물 총 개수를 기준으로 20% 이상의 유전자 전달 효율, 그리고 전달된 유전자를 보유한 식물 총 개수를 기준으로 10% 이상의 표적 유전자 편집 효율을 달성했습니다.
연구팀은 국제 학술지(Plant Science)에 1편을 포함하여 총 5편의 논문과 국내 학술지 4편을 발표했으며, 향후 연구를 지원하기 위해 무기질 성분, 유전자 발현 및 플라스미드 구조 설계 분석 데이터 시스템을 구축했습니다. 또한, 본 프로젝트는 교육 목표도 달성하여 3명의 석사 과정 학생들이 성공적으로 논문 지도 및 심사를 통과함으로써 농업 응용 생명공학 분야의 우수한 인력 양성에 기여했습니다.
실질적인 영향 측면에서 볼 때, 철분과 아연 함량이 높고 카드뮴 함량이 낮은 쌀 품종은 베트남 대부분의 가정에서 주요 에너지원인 쌀의 국가 영양 안보에 상당한 이점을 가져다줄 것으로 기대됩니다. 쌀알에 카드뮴이 축적되는 양이 줄어들면 식품 안전성이 향상되고 장기간 섭취로 인한 카드뮴 노출 위험이 감소합니다.
경제 및 시장 관점에서 영양이 향상된 개량 벼 품종은 베트남 쌀의 위상을 높이고 수출 시장의 엄격한 품질 기준을 충족하며 고부가가치 농업 발전 방향에 부합하는 데 도움이 될 것입니다. 연구팀은 또한 향후 다지역 시험 재배, 생장 및 수확량 평가, 생물 안전성 검증, 그리고 육종 연구를 위한 재료를 연구기관, 대학 및 식물 육종 기업으로 이전하는 것을 다음 단계로 제시했습니다.
본 프로젝트의 성공은 베트남 농업에서 유전자 편집 기술의 큰 잠재력을 입증하며, 미량 영양소가 풍부하고 안전하며 부가가치가 높은 벼 품종 개발을 위한 견고한 과학적 기반을 마련했습니다. 영양 요구량 증가와 벼 품질 기준 강화라는 맥락에서, 이러한 성과는 연구적 의미뿐 아니라 광범위한 적용 가능성을 열어주며, 현대적이고 지속 가능한 지식 기반 농업 발전에 기여할 것입니다.
출처: https://daidoanket.vn/crispr-mo-duong-cho-nong-nghiep-viet-nam.html






![[사진] 제15대 국회 제10차 회기 폐막식](/_next/image?url=https%3A%2F%2Fvphoto.vietnam.vn%2Fthumb%2F1200x675%2Fvietnam%2Fresource%2FIMAGE%2F2025%2F12%2F11%2F1765448959967_image-1437-jpg.webp&w=3840&q=75)























































![[공식] 미사 그룹, 기업, 가정 및 정부를 위한 인공지능(AI) 에이전트 구축 분야에서 선도적인 브랜드 포지셔닝 발표](https://vphoto.vietnam.vn/thumb/402x226/vietnam/resource/IMAGE/2025/12/11/1765444754256_agentic-ai_postfb-scaled.png)




















































댓글 (0)